مثيلة الدنا

مثيلة الدنا (إنگليزية: DNA methylation)، هي عملية حيوية تضاف بواسطتها مجموعة المثيل إلى جزيء الدنا. يمكن أن تغير المثيلة نشاط جزء من الدنا دون تغيير تسلسله. عندما تجدث في محفز الجين، تعمل مثيلة الدنا عادةً على كبت النسخ الجيني. في الثدييات، تعتبر مثيلة الدنا أمرًا ضروريًا للتطور الطبيعي وترتبط بعدد من العمليات الرئيسية بما في ذلك البصمة الجينية، تعطيل الكروموسوم إكس، قمع العنصر القابل للنقل، [[الشيخوخة] ] والتسرطن.
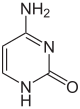
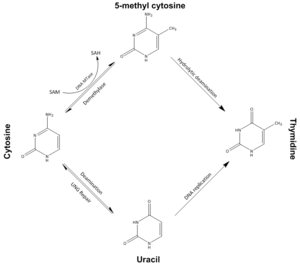
اعتبارًا من عام 2016، تم العثور على قاعدتين نوويتين يحدث عليهما مثيلة الدنا الإنزيمية الطبيعية: الأدنين والسيتوسين. القواعد المعدلة هي N6-methyladenine[1], 5-methylcytosine[2] and N4-methylcytosine.[3]
| القاعدة المعدلة | 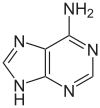
|
|
||||||
| أدنين, A | سيتوسين , C | |||||||
| النماذج المعدلة | 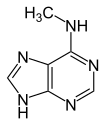
|
|

|
|||||
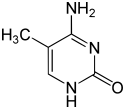
| N6-Methyladenine, 6mA | 5-Methylcytosine, 5mC | N4-Methylcytosine, 4mC | ||||||
الوظيفة المحفوظة لمثيلة الدنا
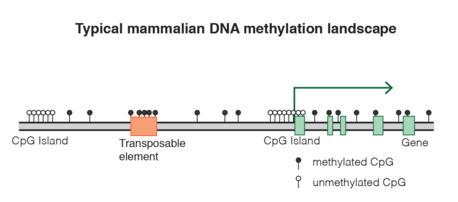
جزر CpG
كبتمعززات كثافة-CpG
كبت العناصر القابلة للنقل
توسع الجينوم
مثيلة الجسم الجيني للجينات عالية النسخ
في الثدييات
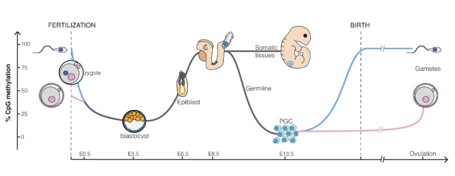
أثناء التطور الجنيني
في السرطان
في العديد من العمليات المرضية، مثل السرطان، تكتسب محفزات الجينات جزر CpG فرط الميثيل غير الطبيعي، مما يؤدي إلى إسكات النسخ الذي يمكن أن ترثه الخلايا الوليدة بعد انقسام الخلايا.[4] تم التعرف على تعديلات مثيلة الدنا كعنصر مهم في تطور السرطان. ينشأ نقص الميثيل، بشكل عام، في وقت مبكر ويرتبط بعدم استقرار الكروموسومات وفقدان البصمة، في حين يرتبط فرط الميثيل بالمحفزات ويمكن أن ينشأ بشكل ثانوي لإسكات الجينات (كابت الجينات الورمية)، ولكنه قد يكون هدفًا العلاج اللاجيني.[5]
يرتبط نقص الميثيل العالمي أيضًا بتطور السرطان من خلال آليات مختلفة.[6] عادة ما يكون فرط ميثيل الجينات الكابتة للورم ونقص ميثيل الجينات الورمية.[7]
بشكل عام، أثناء التقدم إلى السرطان، يتم إسكاتها أو تنشيطها. على الرغم من أن إسكات بعض الجينات في السرطانات يحدث عن طريق الطفرة، إلا أن نسبة كبيرة من إسكات الجينات المسببة للسرطان تكون نتيجة لتغير مثيلة الدنا (انظر مثيلة الدنا في السرطان). تحدث مثيلة الدنا التي تسبب إسكات السرطان عادةً في جزيرة CpG|جزر CpG الموجودة في محفزات جينات ترميز الپروتين.
تؤدي التعبيرات المتغيرة لـ microRNA أيضًا إلى إسكات أو تنشيط العديد من الجينات في تطور السرطان (انظر microRNAs في السرطان). يحدث تعبير microRNA المتغير من خلال فرط/نقص الميثيل من مواقع CpG في جزر CpG في المحفزات المتحكمة في نسخ microRNAs.
يبدو أن إسكات جينات إصلاح الدنا من خلال مثيلة جزر CpG في محفزاتها له أهمية خاصة في تطور السرطان (انظر مثيلة جينات إصلاح الدنا في السرطان).
في تصلب الشرايين
تلعب التعديلات التخلقية مثل مثيلة الدنا دوراً في أمراض القلب والأوعية الدموية، بما في ذلك تصلب الشرايين. في النماذج الحيوانية لتصلب الشرايين، تظهر الأنسجة الوعائية، وكذلك خلايا الدم مثل خلايا الدم وحيدة النواة، نقص الميثيل الشامل مع مناطق جينية محددة من فرط الميثيل. يمكن استخدام تعدد أشكال مثيلة الدنا كمؤشر حيوي مبكر لتصلب الشرايين نظرًا لوجودها قبل ملاحظة الآفات، مما قد يوفر أداة مبكرة للكشف والوقاية من المخاطر.[8]
هناك اثنان من أنواع الخلايا المستهدفة لتعدد أشكال مثيلة الدنا هما الخلايا الوحيدة والخلايا الليمفاوية، والتي تعاني من نقص الميثيل بشكل عام. إحدى الآليات المقترحة وراء نقص الميثيل الشامل هذا هي ارتفاع مستويات الهوموسيستين مما يسبب فرط هوموسيستين الدم، وهو عامل خطر معروف لأمراض القلب والأوعية الدموية. مستويات الپلازما العالية من الهوموسيستين تمنع نقل ميثيل الدنا، والذي يسبب نقص الميثيل. يؤثر نقص ميثيل الدنا على الجينات التي تغير تكاثر خلايا العضلات الملساء، وتسبب خللًا في الخلايا البطانية، وتزيد من وسطاء الالتهابات، وكلها عوامل حاسمة في تكوين آفات تصلب الشرايين.[9] تؤدي المستويات العالية من الهوموسيستين أيضًا إلى فرط ميثيل جزر CpG في المنطقة المحفزة لجين مستقبل الإستروجين-ألفا (ERα)، مما يتسبب في تخفيضه.[10] يحمي مستقبل الإستروجين-ألفا من تصلب الشرايين بسبب عمله كمثبط للنمو، مما يتسبب في بقاء خلايا العضلات الملساء في حالة هادئة.[11] وبالتالي فإن فرط الميثيل لمحفز مستقبل الإستروجين-ألفا يسمح لخلايا العضلات الملساء الداخلية بالتكاثر بشكل مفرط والمساهمة في تطور آفة تصلب الشرايين.[12]
الجين الآخر الذي يعاني من تغير في حالة المثيلة في تصلب الشرايين هو ناقل أحادي الكربوكسيل (MCT3)، الذي ينتج پروتين مسؤول عن نقل اللاكتات والأجسام الكيتونية الأخرى من العديد من أنواع الخلايا، بما في ذلك خلايا العضلات الملساء الوعائية. في مرضى تصلب الشرايين، هناك زيادة في مثيلة جزر CpG في إكسون 2، مما يقلل من تعبير پروتين MCT3. يؤدي تقليل تنظيم MCT3 إلى إضعاف نقل اللاكتات ويزيد بشكل كبير من تكاثر خلايا العضلات الملساء، مما يساهم بشكل أكبر في آفة تصلب الشرايين. أظهرت تجربة خارج العضية الحية باستخدام عامل إزالة الميثيل ديسيتابين (5-aza-2-ديوكسيتيدين) أنها تحفز تعبير MCT3 بطريقة تعتمد على الجرعة، حيث أصبحت جميع مواقع فرط الميثيل في جزيرة إكسون 2 CpG منزوعة الميثيل بعد العلاج. قد يكون هذا بمثابة عامل علاجي جديد لعلاج تصلب الشرايين، على الرغم من عدم إجراء أي دراسات بشرية حتى الآن.[13]
في فشل القلب
بالإضافة إلى تصلب الشرايين الموصوف أعلاه، تحددت تغيرات جينية محددة في فشل القلب البشري. قد يختلف هذا حسب مسببات المرض. على سبيل المثال، في حالة قصور القلب الإقفاري، رُبطت تغيرات مثيلة الدنا بالتغيرات في التعبير الجيني التي قد توجه التعبير الجيني المرتبط بالتغيرات في استقلاب القلب المعروفة بحدوثها.[14] يجب استكشاف أشكال إضافية من قصور القلب (مثل اعتلال عضلة القلب السكري) والأمراض المصاحبة (مثل البدانة) لمعرفة مدى شيوع هذه الآليات. والأمر الأكثر لفتًا للانتباه هو أنه في حالة فشل القلب، ترتبط هذه التغييرات في مثيلة الدنا بالحالة العرقية والاجتماعية والاقتصادية التي تؤثر بشكل أكبر على كيفية تغيير التعبير الجيني،[15] وقد يؤثر على كيفية علاج قصور القلب لدى المريض.
في الشيخوخة
في البشر والثدييات الأخرى، يمكن استخدام مستويات مثيلة الدنا لتقدير عمر الأنسجة وأنواع الخلايا بدقة، مما يشكل ساعة شيخوخة دقيقة.[16]
أظهرت إحدى الدراسات الطولية لألطفال التوائم أنه، بين سن 5 و10 سنوات، كان هناك اختلاف في أنماط المثيلة بسبب التأثيرات البيئية وليس الجينية.[17] هناك خسارة شاملة لمثيلة الدنا أثناء الشيخوخة.[7]
في دراسة قامت بتحليل ميثيلومات الدنا الكاملة للخلايا التائية CD4+ لحديثي الولادة، وفرد يبلغ 26 عامًا وفرد يبلغ 103 عامًا ، لوحظ أن فقدان المثيلة يتناسب مع العمر.[18] لوحظ وجود CpGs ناقصة مثيلة الدنا لدى المعمر مقارنة بحديثي الولادة الذين غطوا جميع الأجزاء الجينومية (المحفزات، المناطق الجينية، البين جينية، والإكسونية).[19] ومع ذلك، فإن بعض الجينات تصبح مفرطة الميثيل مع تقدم العمر، بما في ذلك جينات مستقبلات الاستروجين، p16، وعامل النمو الشبيه بالأنسولين 2.[7]
في التمارين الرياضية
تبين أن التمارين الرياضية عالية الكثافة تؤدي إلى انخفاض مثيلة الدنا في العضلات الهيكلية.[20] بعد التمرين عالي الكثافة تنخف مثيلة PGC-1α ومحفز PDK4 على الفور، في حين أن PPAR-γ لم تنخفض مثيلتها إلى بعد ثلاث ساعات من التمرين.[20] في الوقت نفسه، أدت ستة أشهر من التمارين الرياضية لدى الرجال في منتصف العمر الذين كانوا لا يتمرنون في السابق إلى زيادة المثيلة في الأنسجة الدهنية.[21] أظهرت إحدى الدراسات زيادة محتملة في مثيلة الدنا الجينومي الشامل لخلايا الدم البيضاء مع زيادة النشاط البدني لدى الأشخاص غير ذوي الأصول الإسپانية.[22]
في تمايز الخلايا البائية
أظهرت دراسة بحثت في مثلية الخلايا البائية طوال دورة التمايز الخاصة بهم، باستخدام تسلسل ثنائي كبريتات-الجينومي الكامل (WGBS)، فإن هناك تماثلًا لليثيل من المراحل الأولى إلى المراحل الأكثر تمايزًا. يكون أكبر اختلاف في المثيلة بين مراحل خلايا المركز الجرثومية وخلايا الذاكرة البائية. علاوة على ذلك، أظهرت هذه الدراسة أن هناك تشابهًا بين أورام الخلايا البائية والخلايا البائية طويلة العمر في بصمات مثيلة الحمض النووي.[23]
في المخ
تلخص مراجعتان الأدلة على أن تغيرات مثيلة الدنا في الخلايا العصبية للمخ لها أمية في التعلم والذاكرة.[24][25] يعتبر تكييف الخوف (احد أشكال التعلم الترابطي) في الحيوانات، مثل الفئران والجرذان، سريعًا وقويًا للغاية في تكوين الذكريات.[26] في الفئران[27] وفي الجرذان[28] تكييف الخوف السياقي، خلال 1-24 ساعة، يرتبط بتغيير مثيلات عدة آلاف من السيتوزينات في الدنا في جينات الخلايا العصبية للحـُصين. بعد أربع وعشرين ساعة من تكييف الخوف السياقي، جرى مثيلة 9.2% من الجينات الموجودة في الخلايا العصبية للحـُصين بشكل تفاضلي.[28] في الفئران،[27] عند فحصها بعد أربعة أسابيع من التكييف، أُعيد تعيين مثيلات الحـُصين ونزع مثيلة إلى الظروف الساذجة الأصلية. هناك حاجة إلى الحـُصين لتكوين الذكريات، لكن الذكريات لا تُخزن هناك.
بالنسبة لهذه الفئران، بعد أربعة أسابيع من تكييف الخوف السياقي، حدثت عمليات مثيلة ونزع مثيلة CpG تفاضلية كبيرة في الخلايا العصبية القشرية أثناء صيانة الذاكرة، وكان هناك 1.223 جينًا ميثليًا تفاضليًا في القشرة الحزامية الأمامية للفئران.[27] تلخص هذه الآليات التي توجه مثيلات الدنا الجديدة وإزالة مثيلة الدنا الجديدة في الحـُصين أثناء إنشاء الذاكرة عام 2022.[29] أشارت تلك المراجعة أيضًا إلى الآليات التي أدت من خلالها الأنماط الجديدة من المثيلة إلى ظهور أنماط جديدة من التعبير مرسال الرنا. بعد ذلك نُقلت مراسيل الرنا الجديدة بواسطة جزيئات messenger RNP (الحبيبات العصبية) إلى نقاط الاشتباك العصبي في الخلايا العصبية، حيث يمكن ترجمتها إلى پروتينات.[29] يبدو أن التغييرات النشطة في مثيلة الدنا العصبي وإزالة المثيلة تعمل كوحدات تحكم في القياس التشابكي ومستقبل الگلوتامات في التعلم وتكوين الذاكرة.[24]
ناقلة مثيل الدنا (في الثدييات)
في النباتات
في الحشرات
في الفطريات
في حقيقيات النوى الأخرى
في الجراثيم

الاستنساخ الجزيئي
التحقق
المناطق الممثيلة تفاضلياً (DMRs)
علامات مثيلة الدنا
في تحديد وكشف سوائل الجسم
التنبؤ الحاسوبي
انظر أيضاً
- 5-Hydroxymethylcytosine
- 5-ميثيلسايتوسين
- 7-ميثيلگوانوسين
- الانخفاض في مثيلة الدنا 1 (DDM1)، دين مثيلة نباتي
- عامل نزع المثيلة
- مناطق الميثلة التفاضلية
- نزع مثيلة الدنا
- إعادة برمجة مثيلة الدنا
- علم التخلق، والذي يعتبر مثيل الدنا عاملاً مساهماً هاماً فيها
- ساعة الشيخوخة، طريقة لحساب العمر على أساس مثيلة الدنا
- Epigenome
- الجينوم
- دمغ جينومي، قمع موروث للأليل، يعتمد على مثيلة الدنا
- MethBase قاعدة بيانات مثيلة الدنا المستضافة على UCSC Genome Browser
- MethDB قاعدة بيانات مثيلة الدنا
- N6-مثيلأدينوزين
المصادر
- ^ D. B. Dunn, J. D. Smith: The occurrence of 6-methylaminopurine in deoxyribonucleic acids. In: Biochem J. 68(4), Apr 1958, S. 627–636. PMID 13522672. قالب:PMC.
- ^ B. F. Vanyushin, S. G. Tkacheva, A. N. Belozersky: Rare bases in animal DNA. In: Nature. 225, 1970, S. 948–949. PMID 4391887.
- ^ Melanie Ehrlich, Miguel A. Gama-Sosa, Laura H. Carreira, Lars G. Ljungdahl, Kenneth C. Kuo, Charles W. Gehrke: DNA methylation in thermophilic bacteria: N6-methylcytosine, 5-methylcytosine, and N6-methyladenine. In: Nucleic Acids Research. 13, 1985, S. 1399. PMID 4000939. قالب:PMC.
- ^ Wang YP, Lei QY (May 2018). "Metabolic recoding of epigenetics in cancer". Cancer Communications. 38 (1): 25. doi:10.1186/s40880-018-0302-3. PMC 5993135. PMID 29784032.
{{cite journal}}: CS1 maint: unflagged free DOI (link) - ^ Daura-Oller E, Cabre M, Montero MA, Paternain JL, Romeu A (April 2009). "Specific gene hypomethylation and cancer: new insights into coding region feature trends". Bioinformation. 3 (8): 340–3. doi:10.6026/97320630003340. PMC 2720671. PMID 19707296.
- ^ Craig, JM; Wong, NC (editor) (2011). Epigenetics: A Reference Manual. Caister Academic Press. ISBN 978-1-904455-88-2.
{{cite book}}:|author=has generic name (help)CS1 maint: multiple names: authors list (link) - ^ أ ب ت Gonzalo S (August 2010). "Epigenetic alterations in aging". Journal of Applied Physiology. 109 (2): 586–97. doi:10.1152/japplphysiol.00238.2010. PMC 2928596. PMID 20448029.
- ^ Lund G, Andersson L, Lauria M, Lindholm M, Fraga MF, Villar-Garea A, Ballestar E, Esteller M, Zaina S (July 2004). "DNA methylation polymorphisms precede any histological sign of atherosclerosis in mice lacking apolipoprotein E". The Journal of Biological Chemistry. 279 (28): 29147–54. doi:10.1074/jbc.m403618200. PMID 15131116.
- ^ Castro R, Rivera I, Struys EA, Jansen EE, Ravasco P, Camilo ME, Blom HJ, Jakobs C, Tavares de Almeida I (August 2003). "Increased homocysteine and S-adenosylhomocysteine concentrations and DNA hypomethylation in vascular disease". Clinical Chemistry. 49 (8): 1292–6. doi:10.1373/49.8.1292. PMID 12881445.
- ^ Huang YS, Zhi YF, Wang SR (October 2009). "Hypermethylation of estrogen receptor-alpha gene in atheromatosis patients and its correlation with homocysteine". Pathophysiology. 16 (4): 259–65. doi:10.1016/j.pathophys.2009.02.010. PMID 19285843.
- ^ Dong C, Yoon W, Goldschmidt-Clermont PJ (August 2002). "DNA methylation and atherosclerosis". The Journal of Nutrition. 132 (8 Suppl): 2406S–2409S. doi:10.1093/jn/132.8.2406S. PMID 12163701.
- ^ Ying AK, Hassanain HH, Roos CM, Smiraglia DJ, Issa JJ, Michler RE, Caligiuri M, Plass C, Goldschmidt-Clermont PJ (April 2000). "Methylation of the estrogen receptor-alpha gene promoter is selectively increased in proliferating human aortic smooth muscle cells". Cardiovascular Research. 46 (1): 172–9. doi:10.1016/s0008-6363(00)00004-3. PMID 10727665.
- ^ Zhu S, Goldschmidt-Clermont PJ, Dong C (August 2005). "Inactivation of monocarboxylate transporter MCT3 by DNA methylation in atherosclerosis". Circulation. 112 (9): 1353–61. doi:10.1161/circulationaha.104.519025. PMID 16116050.
- ^ Pepin ME, Ha CM, Crossman DK, Litovsky SH, Varambally S, Barchue JP, Pamboukian SV, Diakos NA, Drakos SG, Pogwizd SM, Wende AR (March 2019). "Genome-wide DNA methylation changes associated with cardiac transcriptional profiles in human ischemic heart failure". Laboratory Investigation. 99 (3): 371–386. doi:10.1038/s41374-018-0104-x. PMC 6515060. PMID 30089854.
- ^ Pepin ME, Ha CM, Potter LA, Bakshi S, Barchue JP, Haj asaad A, Pogwizd SM, Pamboukian SV, Hidalgo BA, Vickers SM, Wende AR (March 2021). "Racial and socioeconomic disparity associates with differences in cardiac DNA methylation among men with end-stage heart failure". American Journal of Physiology. Heart and Circulatory Physiology. Online ahead of print (5): H2066–H2079. doi:10.1152/ajpheart.00036.2021. ISSN 0363-6135. PMC 8163657. PMID 33769919.
- ^ Horvath S (2013). "DNA methylation age of human tissues and cell types". Genome Biology. 14 (10): R115. doi:10.1186/gb-2013-14-10-r115. PMC 4015143. PMID 24138928.
{{cite journal}}: CS1 maint: unflagged free DOI (link) - ^ Wong CC, Caspi A, Williams B, Craig IW, Houts R, Ambler A, Moffitt TE, Mill J (August 2010). "A longitudinal study of epigenetic variation in twins". Epigenetics. 5 (6): 516–26. doi:10.4161/epi.5.6.12226. PMC 3322496. PMID 20505345.
- ^ Heyn, Holger; Li, Ning; Ferreira, Humberto J.; Moran, Sebastian; Pisano, David G.; Gomez, Antonio; Diez, Javier; Sanchez-Mut, Jose V.; Setien, Fernando; Carmona, F. Javier; Puca, Annibale A. (2012-06-26). "Distinct DNA methylomes of newborns and centenarians". Proceedings of the National Academy of Sciences (in الإنجليزية). 109 (26): 10522–10527. Bibcode:2012PNAS..10910522H. doi:10.1073/pnas.1120658109. ISSN 0027-8424. PMC 3387108. PMID 22689993.
- ^ Heyn H, Li N, Ferreira HJ, Moran S, Pisano DG, Gomez A, Diez J, Sanchez-Mut JV, Setien F, Carmona FJ, Puca AA, Sayols S, Pujana MA, Serra-Musach J, Iglesias-Platas I, Formiga F, Fernandez AF, Fraga MF, Heath SC, Valencia A, Gut IG, Wang J, Esteller M (June 2012). "Distinct DNA methylomes of newborns and centenarians". Proceedings of the National Academy of Sciences of the United States of America. 109 (26): 10522–7. Bibcode:2012PNAS..10910522H. doi:10.1073/pnas.1120658109. PMC 3387108. PMID 22689993.
- ^ أ ب Barrès R, Yan J, Egan B, Treebak JT, Rasmussen M, Fritz T, Caidahl K, Krook A, O'Gorman DJ, Zierath JR (March 2012). "Acute exercise remodels promoter methylation in human skeletal muscle". Cell Metabolism. 15 (3): 405–11. doi:10.1016/j.cmet.2012.01.001. PMID 22405075.
- ^ Rönn T, Volkov P, Davegårdh C, Dayeh T, Hall E, Olsson AH, Nilsson E, Tornberg A, Dekker Nitert M, Eriksson KF, Jones HA, Groop L, Ling C (June 2013). "A six months exercise intervention influences the genome-wide DNA methylation pattern in human adipose tissue". PLOS Genetics. 9 (6): e1003572. doi:10.1371/journal.pgen.1003572. PMC 3694844. PMID 23825961.
{{cite journal}}: CS1 maint: unflagged free DOI (link) - ^ Zhang FF, Cardarelli R, Carroll J, Zhang S, Fulda KG, Gonzalez K, Vishwanatha JK, Morabia A, Santella RM (March 2011). "Physical activity and global genomic DNA methylation in a cancer-free population". Epigenetics. 6 (3): 293–9. doi:10.4161/epi.6.3.14378. PMC 3092677. PMID 21178401.
- ^ خطأ استشهاد: وسم
<ref>غير صحيح؛ لا نص تم توفيره للمراجع المسماة:0 - ^ أ ب Sweatt JD (May 2016). "Dynamic DNA methylation controls glutamate receptor trafficking and synaptic scaling". J. Neurochem. 137 (3): 312–30. doi:10.1111/jnc.13564. PMC 4836967. PMID 26849493.
- ^ Kim S, Kaang BK (January 2017). "Epigenetic regulation and chromatin remodeling in learning and memory". Exp. Mol. Med. 49 (1): e281. doi:10.1038/emm.2016.140. PMC 5291841. PMID 28082740.
- ^ Schafe GE, Nadel NV, Sullivan GM, Harris A, LeDoux JE (1999). "Memory consolidation for contextual and auditory fear conditioning is dependent on protein synthesis, PKA, and MAP kinase". Learn. Mem. 6 (2): 97–110. doi:10.1101/lm.6.2.97. PMC 311283. PMID 10327235.
- ^ أ ب ت Halder R, Hennion M, Vidal RO, Shomroni O, Rahman RU, Rajput A, Centeno TP, van Bebber F, Capece V, Garcia Vizcaino JC, Schuetz AL, Burkhardt S, Benito E, Navarro Sala M, Javan SB, Haass C, Schmid B, Fischer A, Bonn S (January 2016). "DNA methylation changes in plasticity genes accompany the formation and maintenance of memory". Nat. Neurosci. 19 (1): 102–10. doi:10.1038/nn.4194. PMC 4700510. PMID 26656643.
- ^ أ ب Duke CG, Kennedy AJ, Gavin CF, Day JJ, Sweatt JD (July 2017). "Experience-dependent epigenomic reorganization in the hippocampus". Learn. Mem. 24 (7): 278–288. doi:10.1101/lm.045112.117. PMC 5473107. PMID 28620075.
- ^ أ ب Bernstein C (2022). "DNA Methylation and Establishing Memory". Epigenet Insights. 15: 25168657211072499. doi:10.1177/25168657211072499. PMC 8793415. PMID 35098021.
- ^ Matthew J. Blow, Tyson A. Clark, Chris G. Daum, Adam M. Deutschbauer, Alexey Fomenkov, Roxanne Fries, Jeff Froula, Dongwan D. Kang, Rex R. Malmstrom, Richard D. Morgan, Janos Posfai, Kanwar Singh, Axel Visel, Kelly Wetmore, Zhiying Zhao, Edward M. Rubin, Jonas Korlach, Len A. Pennacchio, Richard J. Roberts: The Epigenomic Landscape of Prokaryotes. In: PLoS Genet. 12(2), Feb 2016, S. e1005854. doi:10.1371/journal.pgen.1005854. PMID 26870957. PMC 4752239
قراءات إضافية
- Law JA, Jacobsen SE (March 2010). "Establishing, maintaining and modifying DNA methylation patterns in plants and animals". Nature Reviews Genetics. 11 (3): 204–20. doi:10.1038/nrg2719. PMC 3034103. PMID 20142834.
- Straussman R, Nejman D, Roberts D, Steinfeld I, Blum B, Benvenisty N, Simon I, Yakhini Z, Cedar H (May 2009). "Developmental programming of CpG island methylation profiles in the human genome". Nature Structural & Molecular Biology. 16 (5): 564–71. doi:10.1038/nsmb.1594. PMID 19377480. S2CID 8804930.
- Patra SK (April 2008). "Ras regulation of DNA-methylation and cancer". Experimental Cell Research. 314 (6): 1193–201. doi:10.1016/j.yexcr.2008.01.012. PMID 18282569.
- Patra SK, Patra A, Rizzi F, Ghosh TC, Bettuzzi S (June 2008). "Demethylation of (Cytosine-5-C-methyl) DNA and regulation of transcription in the epigenetic pathways of cancer development". Cancer and Metastasis Reviews. 27 (2): 315–34. doi:10.1007/s10555-008-9118-y. hdl:11381/1797001. PMID 18246412. S2CID 22435914.
وصلات خارجية
- MeSH DNA+Methylation
- ENCODE threads explorer Non-coding RNA characterization. Nature (journal)
- PCMdb Pancreatic Cancer Methylation Database.
- Nagpal G, Sharma M, Kumar S, Chaudhary K, Gupta S, Gautam A, Raghava GP (February 2014). "PCMdb: pancreatic cancer methylation database". Scientific Reports. 4: 4197. Bibcode:2014NatSR...4E4197N. doi:10.1038/srep04197. PMC 3935225. PMID 24569397.
- SMART Specific Methylation Analysis and Report Tool
- Human Methylation Mark Atlas
- DiseaseMeth Human disease methylation database
- EWAS Atlas A knowledgebase of epigenome-wide association studies