جين قافز

الجين القافز أو الينقول (بالإنگليزية: Transposable element، TE, transposon، أو jumping gene)، هو تسلسل الحمض النووي الذي يمكنه تغيير موقعه داخل الجينوم، وأحياناً تخليق أو عكس الطفرات وتغيير الهوية الجينية للخلية وحجم الجينوم.[1] غالبًا ما يؤدي النقل إلى ازدواجية نفس المادة الجينية. باكتشافها الجينات القافزة، فازت بربرا مكلينتوك بجائزة نوبل في الطب عام 1983.[2]
تشكل الجينات القافزة جزءًا كبيرًا من الجينوم وهي مسؤولة عن الكثير من كتلة الحمض النووي في خلايا حقيقيات النوى. على الرغم من أن الجينات القافزة هي عنصر وراثي أناني، إلا أن العديد منها هام في عمل الجينوم والتطور.[3] الجينات القافزة بالغة الأهمية للباحثين كوسائل لتبديل الحمض النووي داخل العضيات الحية.
هناك فئتان على الأقل من الجينات القافزة: الفئة الأولى من الجينات القافزة وهي هي الجينات القافزة الراجعة (retrotransposon أو تعمل بشكل عام عبر النسخ العكسي، بينما تشفر الفئة الثانية من الجينات القافزة أو جينات الدنا القافزة الپروتين ترانسپوزاز، المطلوب للإدخال والاستئصال، وبعض هذه الجينات القافزة تشفر أيضًا پروتينات أخرى.[4]
اكتشافه
اكتشفت بربرا مكلينتوك أول الجينات القافزة في الذرة الصفراء في معمل كولد سپرنگ هاربر بنيويورك. كانت مكلينتوك تجري تجاربها على نباتات الذرة الصفراء التي كُسرت كروسوماتها.[5]
في شتاء 1944-1945، زرعت مكلينتوك حبات الذرة التي كانت ملقحة ذاتياً، مما يعني أن شعيرات الزهرة تتلقى حبوب اللقاح من بعضها البعض.[5] جاءت هذه الحبات من سلسلة طويلة من النباتات التي تم تلقيحها ذاتيًا، مما تسبب في كسر الأذرع في نهاية الكروموسوم التاسع.[5] بعدما بدأت الذرى في النمو، لاحظت مكلينتوك نمط لوني غير طبيعي على الأوراق.[5] على سبيل المثال، تحتوي ورقة واحدة على اثنتين من البقع المهقاء ذات الحجم المتطابق تقريبًا، وتقع جنبًا إلى جنب على الورقة.[5] افترضت مكلينتوك أنه أثناء الانقسام الخلوي، فقدت بعض الخلايا المادة الجينية، بينما اكتسبت خلايا أخرى ما فقدته.[6] ومع ذلك، عند مقارنة كروموسومات الجيل الحالي من النباتات بالجيل الأم، وجدت أن أجزاء معينة من الكروموسوم قد تغير موقعها.[6] دحض هذا النظرية الجينية الشائعة في ذلك الوقت بأن الجينات مثبتة في موقعها على الكروموسوم. وجدت مكلينتوك أن الجينات لا يمكن أن تتحرك فقط ولكن يمكن أيضًا تشغيلها أو إيقاف تشغيلها لظروف بيئية معينة أو خلال مراحل مختلفة من تطور الخلية.[6]
أظهرت مكلينتوك أيضًا أن الطفرات الجينية يمكن عكسها.[7] عام 1951 قدمت تقريرها عن النتائج التي توصلت إليها، ونشرت مقالًا عن اكتشافاتها في "علم الوراثة" في نوفمبر 1953 بعنوان "تحريض عدم الاستقرار في مناطق مختارة في الذرة الصفراء".[8]
عام 1951 في ندوة كولد سپرنگ هاربر حيث نشرت لأول مرة النتائج التي توصلت إليها، قوبل حديثها بصمت تام.[9] رُفض عملها وقوبلت بالتجاهل إلى حد كبير حتى أواخر الستينيات والسبعينيات عندما اكتشفت الجينات القافزة في البكتيريا بعد اكتشاف مكلينتوك.[10] حازت جائزة نوبل في الطب عام 1983 من أجل اكتشافها الجينات القافزة، بعد أكثر من ثلاثين عاماً من أبحاثها الأولية.[11]
التصنيف
الجينات القافزة الراجعة
جينات الدنا القافزة
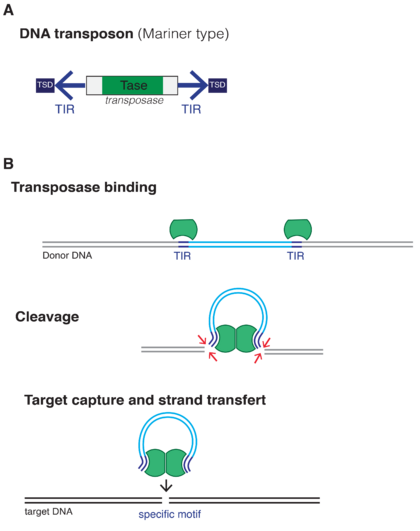
B. آلية النقل: Two transposases recognize and bind to TIR sequences, join and promote DNA double-strand cleavage. The DNA-transposase complex then inserts its DNA cargo at specific DNA motifs elsewhere in the genome, creating short TSDs upon integration.[12]
المستقلة والغير مستقلة
الفئة الثالثة
التوزيع
أمثلة
الآثار السلبية
آليات الطفرات
الأمراض
الأمراض التي عادة ما تسببه الجينات القافزة:
- الهيموفيليا "أ" و"ب"
- LINE1 (L1) ثبت أن الجينات القافزة التي تتموضع على العامل الثامن البشري تسبب الهيموفيليا. [13]
- نقص المناعة المشترك الحاد
- إدخال L1 في السليلة القولونية الغدية يسبب سرطان القولون، وقد تأكد أن الجينات القافزة تلعب دوراً هاماً في تطور المرض.[14]
- الپورفيريا
- إدخال جين Alu إلى جين PBGD يؤدي إلى التداخل مع منطقة التشفير ويؤدي إلى الپورفيريا الحادة المتقطعة [15] (AIP).
- الاستعداد للإصابة بالسرطان
- LINE1(L1) ترتبط الجينات القافزة والجينات القافزة الراجعة الأخرى بالسرطان لأنها تسبب عدم الاستقرار الجينومي.[13]
- الحثل العضلي الدوشيني.[16][17]
- يسببه إدخال جين SVA القافز في جين فوكوتين (FKTN) الذي يجعل الجين غير نشط.[13]
- مرض ألزايمر وأمراض تاوية أخرى
- يمكن أن يتسبب عدم انتظام الجينات القافزة في موت الخلايا العصبية، مما يؤدي إلى اضطرابات التنكس العصبي [18]
معدل الانتقال، الاستقراء والدفاع
التطور
الدافع التطوري للجينات القافزة في السياق الجيني
التطبيقات
الهندسة الوراثية
- تستخدم الطفرات الإدراجية ميزات الجينات القافزة لإدخال التسلسل. في معظم الحالات، يستخدم هذا لإزالة تسلسل الحمض النووي أو التسبب في حدوث طفرة انزياح الإطار frameshift mutation.
- في بعض الحالات، يمكن أن يؤدي إدخال الجينات القافزة في الجين إلى تعطيل وظيفة هذا الجين بطريقة قابلة للعكس حيث يؤدي الاستئصال بوساطة الترانسوپوزاز من جينات الدنا القافزة إلى استعادة وظيفة الجين.
- ينتج عن هذا نباتات تمتلك فيها الخلايا المجاورة أنماط جينية مختلفة.
- تسمح هذه الميزة للباحثين بالتمييز بين الجينات التي يجب أن تكون موجودة داخل الخلية لتعمل (ذاتية الخلية) والجينات التي تنتج تأثيرات ملحوظة في الخلايا غير تلك التي يتم فيها التعبير عن الجين.
الأداة الجينية
بالإضافة إلى الصفات المذكورة في الهندسة الوراثية، تعتبر الجينات الوراثية أداة وراثية أيضاً:
- تستخدم الجينات القافزة لتحليل التعبير الجيني وعمل الپروتين في signature-tagging mutagenesis.
- تتيح هذه الأداة التحليلية للباحثين القدرة على تحديد تعبير النمط الظاهري للتسلسل الجيني. كما تعمل هذه التقنية التحليلية على تغيير موضع الاهتمام المطلوب بحيث يمكن مقارنة الأنماط الظاهرية للجين الأصلي والجين المتحور.
تطبيقات خاصة
- تعتبر الجينات القافزة أيضًا أداة مستخدمة على نطاق واسع لطفرات معظم العضيات القابلة للتجربة. تم استخدام نظام الجينات القافزة الجمال النائم على نطاق واسع كعلامة إدخال لتحديد جينات السرطان.[19]
- فئة Tc1/مارينر-من الجينات القافزة في نظام الجمال النائم، حازت جائزة جزيء العام 2009،[20] وهي نشطة في خلايا الثدييات وتُفحص لاستخدامها في العلاج الجيني البشري.[21][22][23]
- تستخدم الجينات القافزة في إعادة بناء علم الوراثة العرقي بوسائل التحاليل الحاضرة/الغائبة.[24] يمكن أن تعمل الجينات القافزة كمطفر حيوية في الجراثيم.
- العضيات الشائعة التي تم تطوير استخدام الجينات القافزة فيها بشكل جيد:
تحديد هوية تكرار دو نوڤو
الجينات القافزة المتكيفة
الجينات القافزة في شبكات التحكم الجيني
انظر أيضاً
الهوامش
- Kidwell MG (2005). "Transposable elements". In T.R. Gregory (ed.). The Evolution of the Genome. San Diego: Elsevier. pp. 165–221. ISBN 978-0-123-01463-4.
- Craig NL, Craigie R, Gellert M, and Lambowitz AM, eds. (2002). Mobile DNA II. Washington, DC: ASM Press. ISBN 978-1-555-81209-6.
- Lewin B (2000). Genes VII. Oxford University Press. ISBN 978-0-198-79276-5.
المصادر
- ^ Bourque G, Burns KH, Gehring M, Gorbunova V, Seluanov A, Hammell M, et al. (November 2018). "Ten things you should know about transposable elements". Genome Biology. 19 (1): 199. doi:10.1186/s13059-018-1577-z. PMC 6240941. PMID 30454069.
{{cite journal}}: CS1 maint: unflagged free DOI (link) - ^ McClintock B (June 1950). "The origin and behavior of mutable loci in maize". Proceedings of the National Academy of Sciences of the United States of America. 36 (6): 344–55. Bibcode:1950PNAS...36..344M. doi:10.1073/pnas.36.6.344. PMC 1063197. PMID 15430309.
- ^ Bucher E, Reinders J, Mirouze M (November 2012). "Epigenetic control of transposon transcription and mobility in Arabidopsis". Current Opinion in Plant Biology. 15 (5): 503–10. doi:10.1016/j.pbi.2012.08.006. PMID 22940592.
- ^ Pray LA (2008). "Transposons: The jumping genes". Nature Education. 1 (1): 204.
- ^ أ ب ت ث ج McGrayne SB (1998). Nobel Prize Women in Science: Their Lives, Struggles, and Momentous Discoveries (2nd ed.). Carol Publishing. p. 165. ISBN 978-0-9702256-0-3.
- ^ أ ب ت قالب:Harnvb
- ^ قالب:Harnvb
- ^ McClintock B (November 1953). "Induction of Instability at Selected Loci in Maize". Genetics. 38 (6): 579–99. doi:10.1093/genetics/38.6.579. PMC 1209627. PMID 17247459.
- ^ Ravindran, S. (2012). "Proceedings of the National Academy of Sciences Dec 2012, 109 (50) 20198-20199; DOI: 10.1073/pnas.1219372109". Proceedings of the National Academy of Sciences of the United States of America. 109 (50): 20198–20199. doi:10.1073/pnas.1219372109. PMC 3528533. PMID 23236127.
- ^ Des Jardins J (2010). The Madame Curie Complex: The Hidden History of Women in Science. Feminist Press at CUNY. p. 246. ISBN 978-1-55861-655-4.
- ^ Fedoroff N, Botstein D, eds. (1 January 1992). The Dynamic Genome: Barbara McClintock's Ideas in the Century of Genetics. Cold Spring Harbor Laboratory Press. p. 2. ISBN 978-0-87969-422-7.
- ^ Walter M (2016). Transposon regulation upon dynamic loss of DNA methylation (Thesis). Université Pierre et Marie Curie. doi:10.13140/rg.2.2.18747.21286.
- ^ أ ب ت Kazazian HH, Wong C, Youssoufian H, Scott AF, Phillips DG, Antonarakis SE (March 1988). "Haemophilia A resulting from de novo insertion of L1 sequences represents a novel mechanism for mutation in man". Nature. 332 (6160): 164–6. Bibcode:1988Natur.332..164K. doi:10.1038/332164a0. PMID 2831458.
- ^ Miki Y, Nishisho I, Horii A, Miyoshi Y, Utsunomiya J, Kinzler KW, Vogelstein B, Nakamura Y (February 1992). "Disruption of the APC gene by a retrotransposal insertion of L1 sequence in a colon cancer". Cancer Research. 52 (3): 643–5. PMID 1310068.
- ^ Mustajoki S, Ahola H, Mustajoki P, Kauppinen R (June 1999). "Insertion of Alu element responsible for acute intermittent porphyria". Human Mutation. 13 (6): 431–8. doi:10.1002/(sici)1098-1004(1999)13:6<431::aid-humu2>3.0.co;2-y. PMID 10408772.
- ^ Kazazian HH, Goodier JL (August 2002). "LINE drive. retrotransposition and genome instability". Cell. 110 (3): 277–80. doi:10.1016/S0092-8674(02)00868-1. PMID 12176313.
- ^ Kapitonov VV, Pavlicek A, Jurka J (2006). Anthology of Human Repetitive DNA. Encyclopedia of Molecular Cell Biology and Molecular Medicine. doi:10.1002/3527600906.mcb.200300166. ISBN 978-3527600908.
- ^ Sun W, Samimi H, Gamez M, Zare H, Frost B (August 2018). "Pathogenic tau-induced piRNA depletion promotes neuronal death through transposable element dysregulation in neurodegenerative tauopathies". Nature Neuroscience. 21 (8): 1038–1048. doi:10.1038/s41593-018-0194-1. PMC 6095477. PMID 30038280.
- ^ خطأ استشهاد: وسم
<ref>غير صحيح؛ لا نص تم توفيره للمراجع المسماة:1 - ^ خطأ استشهاد: وسم
<ref>غير صحيح؛ لا نص تم توفيره للمراجع المسماةWojciech Makałowski p. 337-359 - ^ خطأ استشهاد: وسم
<ref>غير صحيح؛ لا نص تم توفيره للمراجع المسماة:2 - ^ خطأ استشهاد: وسم
<ref>غير صحيح؛ لا نص تم توفيره للمراجع المسماة:3 - ^ خطأ استشهاد: وسم
<ref>غير صحيح؛ لا نص تم توفيره للمراجع المسماة:4 - ^ خطأ استشهاد: وسم
<ref>غير صحيح؛ لا نص تم توفيره للمراجع المسماة:5 - ^ Tempel S, Rousseau C, Tahi F, Nicolas J (September 2010). "ModuleOrganizer: detecting modules in families of transposable elements". BMC Bioinformatics. 11: 474. doi:10.1186/1471-2105-11-474. PMC 2955051. PMID 20860790.
{{cite journal}}: CS1 maint: unflagged free DOI (link) - ^ خطأ استشهاد: وسم
<ref>غير صحيح؛ لا نص تم توفيره للمراجع المسماةMobilization of transposons by a mu
وصلات خارجية
- "An immune system so versatile it might kill you". New Scientist (2556). 21 June 2006. – A possible connection between aberrant reinsertions and lymphoma.
- Repbase – a database of transposable element sequences
- RepeatMasker – a computer program used by computational biologists to annotate transposons in DNA sequences
- Use of the Sleeping Beauty Transposon System for Stable Gene Expression in Mouse Embryonic Stem Cells
- Introduction to Transposons, 2018 YouTube video